Continuiamo la serie su GGPLOT2 analizzando la penultima componente, le coordinate.
Le funzioni di questa componente ci permetteranno di modificare alcune caratteristiche degli assi, alcune delle quali sono fondamentali per creare finalmente un pieplot con GGPLOT2.
Ratio e flip degli assi
Partiamo vedendo una funzione che ci permette di decidere in che rapporto (ratio) devono essere gli assi; in altre parole, a quante unità sull’asse y corrisponde una unità dell’asse x? Vediamone subito un esempio.
Figura 1: Grafico di default
Di default, ggplot2 cerca di rendere il grafico più “bello” possibile in quanto a dimensioni degli assi; spesso questo si traduce nell’utilizzo di una scala diversa per i due assi (come in figura 1).
Se invece noi volessimo avere i due assi aventi la stessa scala, dovremmo aggiungere il comando coord_fixed. Il risultato sarebbe questo:
Figura 2: Grafico con coord_fixed
Ora i due assi hanno la stessa scala, e infatti l’asse y è molto schiacciato rispetto all’asse x. Per modificare il rapporto tra l’asse y e l’asse x, e dunque sistemare il grafico, c’è da impostare l’input ratio.
Facendo una prova con ratio = 10, otteniamo questo grafico:
Figura 3: Grafico con ratio settata
Ora il grafico è tornato ad essre chiaro.
Questa funzione può non sembrare molto utile ora, ma potrebbe riverlarlasi nel momento in cui abbiamo bisogno di creare un grafico che sia un po’ schiacciato verso l’asse x o verso l’asse y.
Per concludere questo mini capitolo, vi parlo velocemente della funzione coord_flip che, come potrete intuire dal nome, inverte asse x e asse y. Questo può tornare utile nel momento in cui per necessità siete costretti ad utilizzare geometrie o statistiche sull’asse x (o y) ma vorreste avere un grafico in cui in realtà i valori modificati siano sull’asse y (o viceversa). Vi basterà aggiungere questa piccola funzione alla fine del comando del grafico ed il gioco sarà fatto.
Zoom e trasformazioni
Una cosa che ci permette di fare la componente coord_ è quella di zoomare il grafico senza perdere dei dati (cosa che accadrebbe se settassimo i limiti degli assi usando scales_ in modo tale da fare uno zoom sul grafico).
Il modo per zoomare è dato dagli input xlim e ylim di ogni comando coord_; se nel grafico si è scelto di non usare alcuna modifica coord, bisogna ricordarsi che il valore di default è coord_cartesian e che basterà aggiungere quel comando con gli input giusti.
Nel nostro caso, volendo zoomare il grafico in figura 1 per evidenziare i valori bassi, dovremmo aggiungere coord_cartesian(xlim = c(10, 17), ylim = c(2.5, 4)). Ecco il risultato:
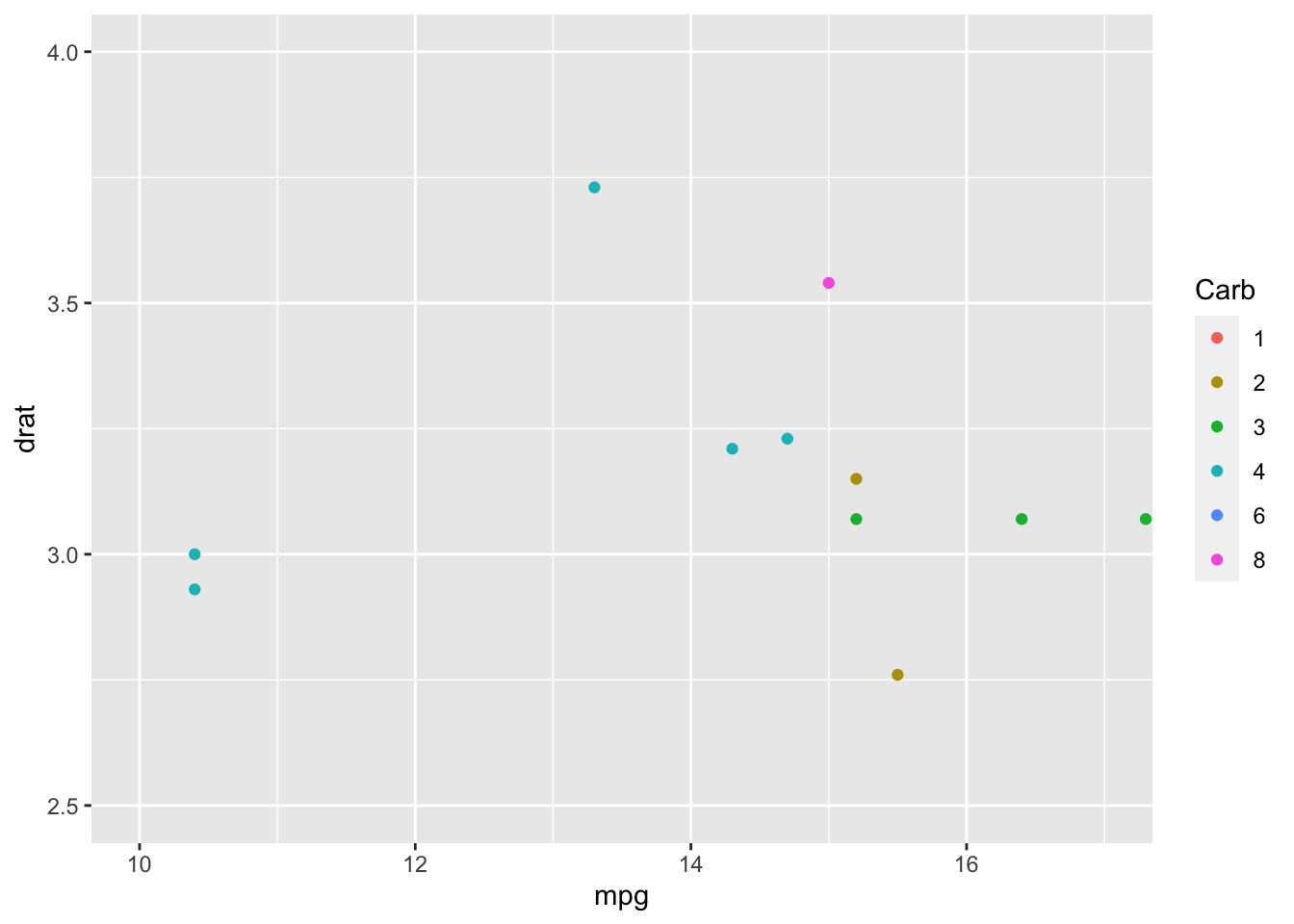
Figura 4: Zoom sulla porzione inferiore del grafico in figura 1
Un discorso molto simile vale per le trasformazioni legate agli assi, come per esempio l’applicazione di una scala logaritmica su un asse.
Il comando è coord_trans, e l’input in questo caso è y (o x) = "log".
Ma perchè queste differenze?
Scale applica i cambiamenti all’inizio, mentre coord lo fa dopo, una volta che i dati sono già plottati. Infatti, settando dei limiti restrittivi con scale, R dirà che ha eliminato dei dati, perchè quei dati non rientrano nei limiti imposti; con coord invece i dati vengono prima plottati tutti, poi viene fatto uno zoom nella sezione delimitata dai limitti settati.
Una cosa simile avviane per le trasformazioni: usando coord per le trasformazioni, i breaks avranno valori più chiari rispetto a quelli creati da scale.
Coordinate polari
Eccoci arrivati ad un comando molto importante per la costruzione di un pieplot (ma non solo!):coord_polar.Questo comando ci permette di trasformare gli assi da cartesiani a polari. Ci sono vari input:
Vediamo subito un esempio in cui sono i dati in y ad essere usati come valori degli angoli:
ggplot(mtcars) +
geom_bar(aes(x = factor(cyl)), fill = c("red3", "blue3", "green4")) +
coord_polar(theta = "y", start = - 1.57) +
expand_limits(y = 15) +
xlab("Cyl") +
scale_y_continuous(breaks = seq(0, 15, 3))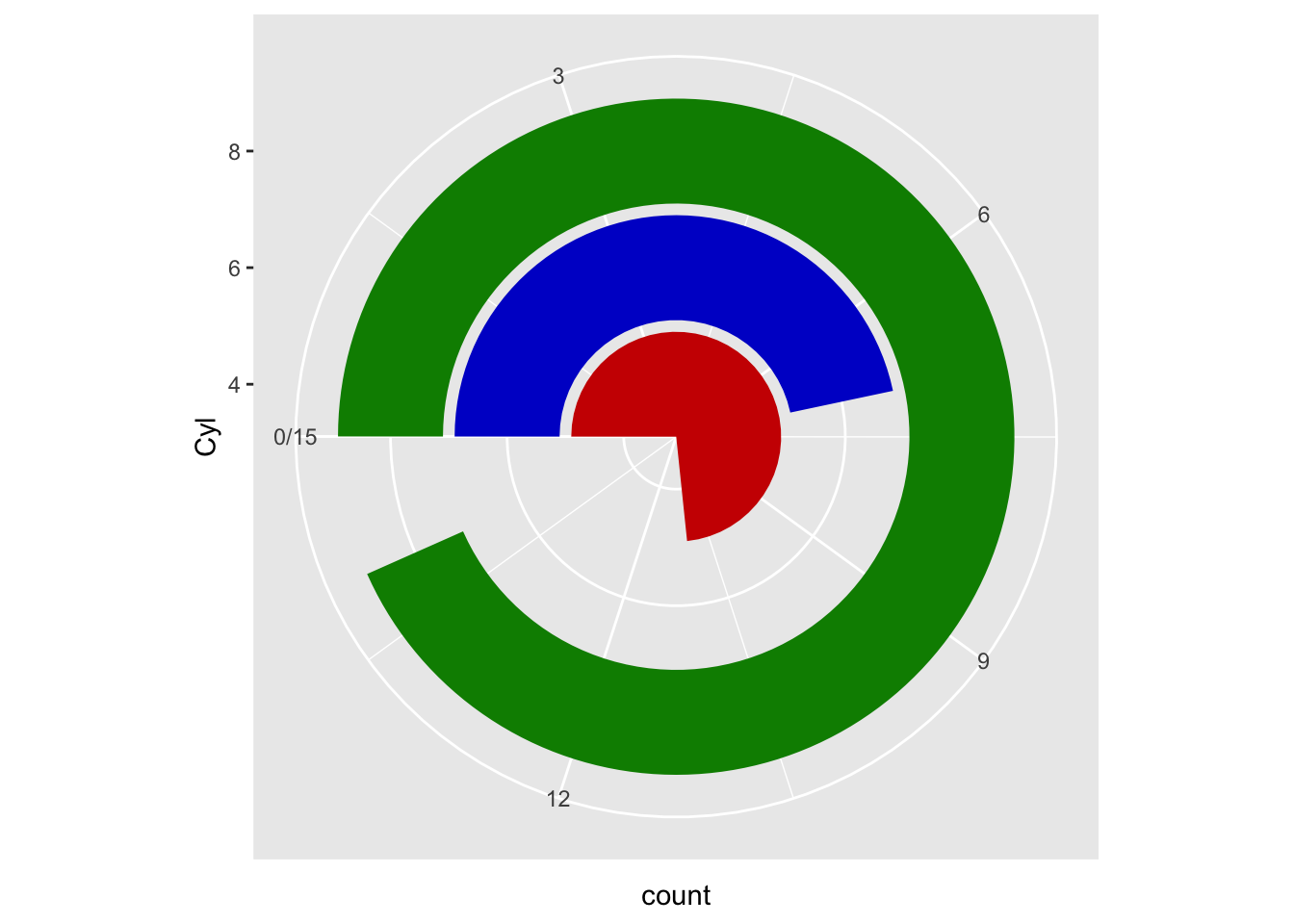
Figura 5: Coordinate polari su un barplot
Ho aggiunto expand_limit perchè di default il limite dell’angolo theta è uguale al valore massimo del corrispettivo asse y delle coordinate cartesiane, facendo diventare un cerchio unico quello relativo agli 8 cilindri.
Pieplot
Vediamo ora come creare un pieplot su ggplot2.
Partiamo dall’esempio in figura 5, stavolta mettendo l’asse x come variabile di angolo teta:
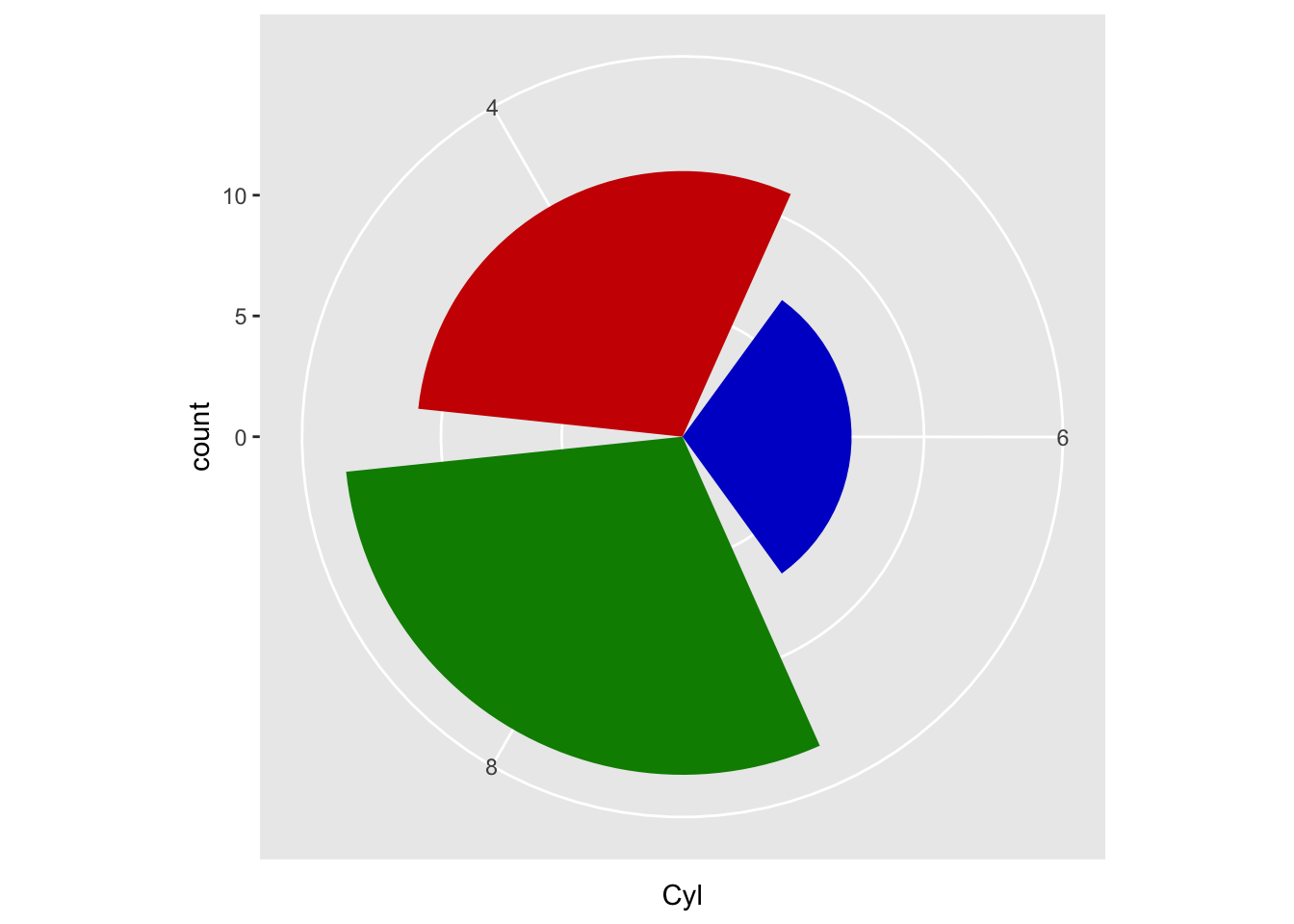
Figura 6: Coord_polar con theta = asse x
Sembra che ci siamo quasi, che sia la direzione giusta per un pieplot. Purtroppo non è così.
Per creare un pieplot su ggplot2 bisogna creare un barplot da un’unica colonna i cui valori sull’asse y rappresentanto i vari valori di riferimento.
# creo un df che racchiuda i valory di cyl e il corrispettivo conteggio
df <- mtcars %>%
group_by(cyl = factor(cyl)) %>%
summarise(n = n())
ggplot(df) +
geom_col(aes(x = 0, y = n, fill = cyl))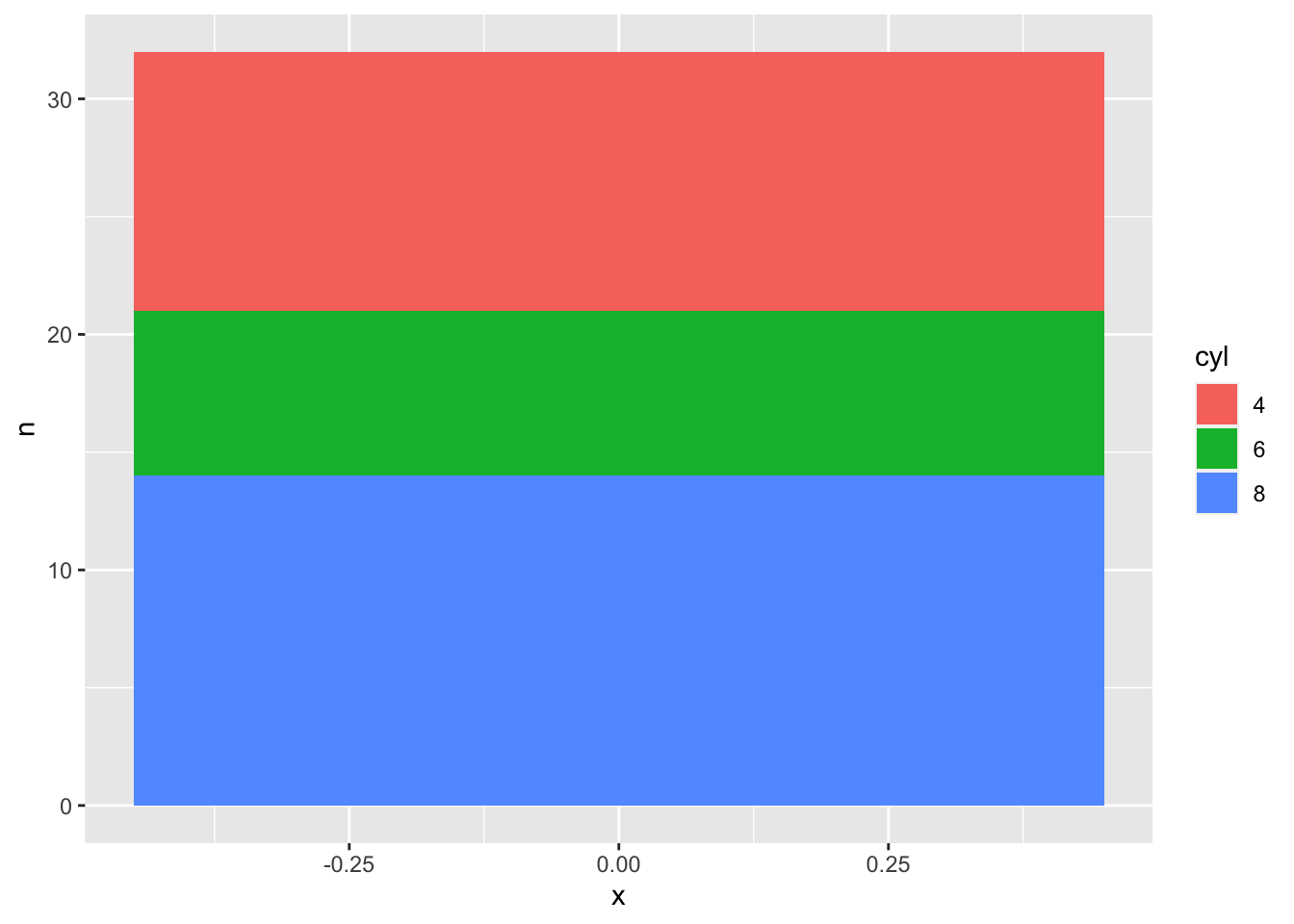
Figura 7: Primo step per pieplot
Una volta arrivati a questo punto, potrebbe venire intuitivo usare i valori in y come valori per l’angolo theta.
ggplot(df) +
geom_col(aes(x = 0, y = n, fill = cyl)) +
coord_polar(theta = "y") +
theme_void()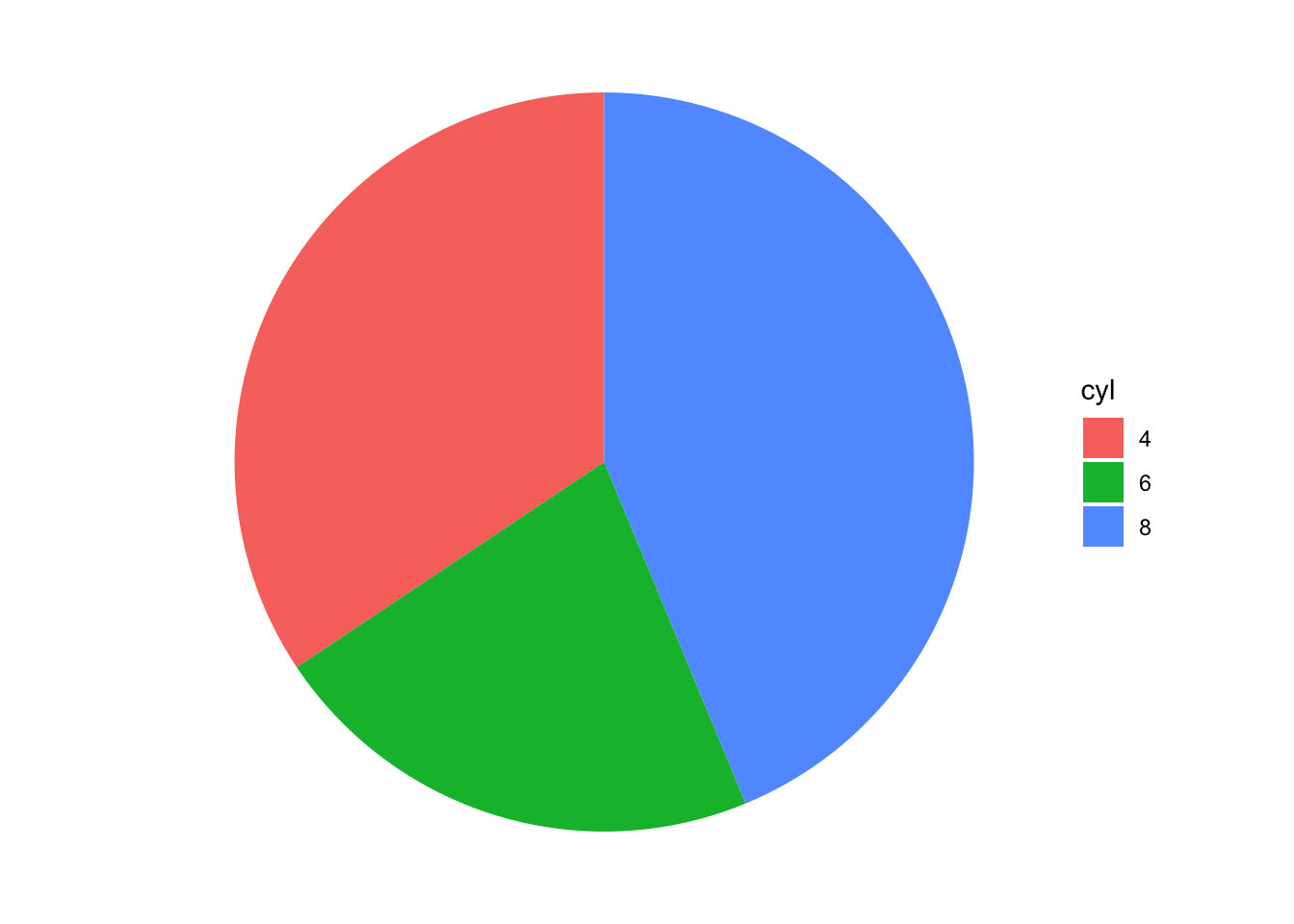
Figura 8: Pieplot completo
Ecco il nostro pieplot.
Tramite geom_text potremmo aggiungere le percentuali e avere un grafico come questo:
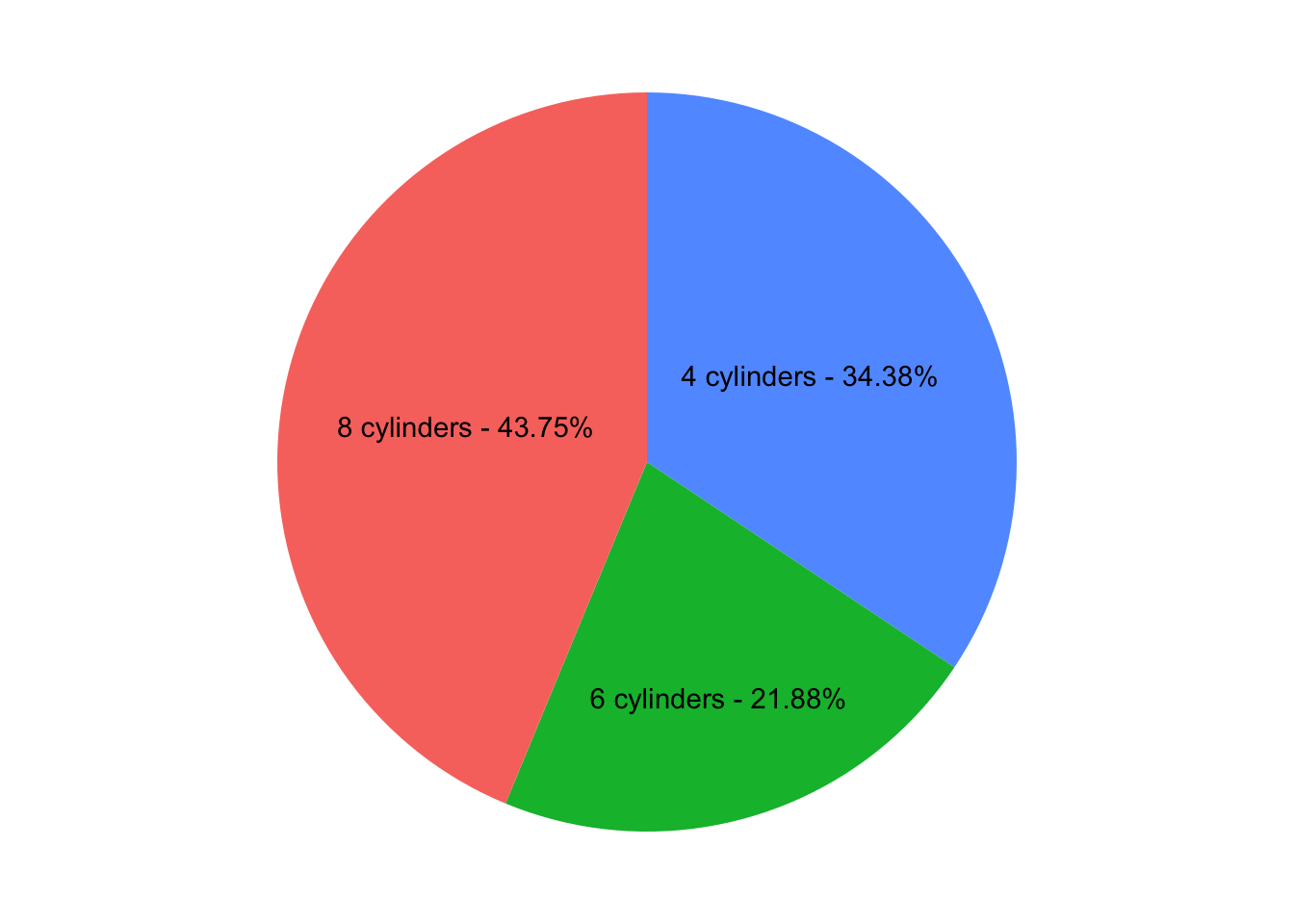
Figura 9: Pieplot con percentuali
Ma vedremo più avanti come utilizzare bene geom_text per annotare informazioni importanti sui nostri grafici.
